Antonio M. Rodríguez García, Universidad de Castilla-La Mancha; Antonio de la Hoz Ayuso, Universidad de Castilla-La Mancha y Enrique Díez Barra, Universidad de Castilla-La Mancha
Las proteínas, esas conocidas moléculas compuestas por secuencias de aminoácidos, están presentes en nuestra alimentación cotidiana y son muy importantes en todos los procesos de la vida. Las combinaciones de los 20 aminoácidos esenciales –desde decenas a miles– generan todas ellas.
El estudio de su composición, función y estructura recibe la atención de los investigadores y de la de Academia Sueca de Ciencias desde hace mucho tiempo: lo han reconocido en los premios Nobel de 1957, 1962, 1972, 1982, 1983, 2004, 2012 y, ahora, 2024.
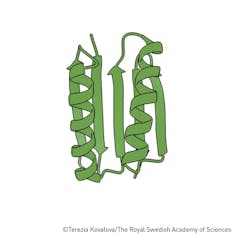 La proteína artificial Top7, diseñada por Brian Kuhlman y Gautam Dantas en el laboratorio de David Baker en la Universidad de Washington.
Terezia Kovalova/The Royal Swedish Academy of Sciences
La proteína artificial Top7, diseñada por Brian Kuhlman y Gautam Dantas en el laboratorio de David Baker en la Universidad de Washington.
Terezia Kovalova/The Royal Swedish Academy of Sciences
¿Será este el último premio Nobel relacionado con las proteínas? Muy probablemente, ya que la contribución de David Baker, Demis Hassabis y John M. Jumper, los galardonados de este año, ha resuelto de un plumazo dos de los grandes retos de la bioquímica.
Por un lado, ha permitido conocer la estructura tridimensional de unas 200 000 proteínas –prácticamente, todas las conocidas– con ayuda de la inteligencia artificial. Por otro, hace posible “crear” proteínas, esto es, identificar la secuencia de aminoácidos que forma una proteína con la estructura tridimensional deseada.
Deconstruyendo los ladrillos de la vida
¿Y para qué interesa conocer la estructura tridimensional de las proteínas y tener la capacidad de crearlas a demanda? Para el beneficio de la humanidad, como reclamaba Alfred Nobel en el testamento que da origen a estos galardones.
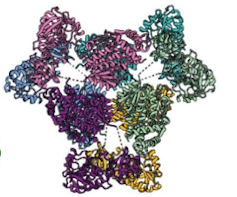 Una enzima (una proteina) bacteriana que causa la resistencia a antibióticos.
Terezia Kovalova/The Royal Swedish Academy of Sciences
Una enzima (una proteina) bacteriana que causa la resistencia a antibióticos.
Terezia Kovalova/The Royal Swedish Academy of Sciences
Saber por qué se produce la resistencia a antibióticos, cuál es el mecanismo para que se desarrollen determinadas enfermedades, entender cómo algunas bacterias descomponen material plástico, acelerar los procedimientos para crear vacunas, crear fármacos específicos o personalizados o nuevos nanomateriales, es, tras las aportaciones de los galardonados, mucho más fácil, rápido y accesible al conjunto de los investigadores de diversos campos.
Para poder construir desde cero
David Baker es especialista en desarrollar programas de química computacional para, estableciendo la premisa de la estructura 3D deseada, identificar qué aminoácidos componen la estructura primaria de la proteína.
En las propias palabras de Baker: “Si quieres construir un avión, no empiezas modificando un pájaro, sino que comprendes los primeros principios de la aerodinámica y construyes máquinas voladoras a partir de ellos”. Esto cambió ya en 2003 la estrategia antes basada en la modificación de proteínas conocidas.
Anuncio del Premio Nobel de Química por el profesor Hans Ellegren, secretario general de la Real Academia Sueca de Ciencias, el 9 de octubre de 2024.
Por su parte Demis Hassabis y John Jumper, este con menos de 40 años, han aprovechado los desarrollos de la inteligencia artificial inicialmente aplicados en juegos de mesa.
El programa Deep Blue consiguió derrotar al campeón del mundo de ajedrez Gary Kasparov en 1997 y después se retiró, ya que no era ese su objetivo final. De la misma manera Alpha Go consiguió derrotar al campeón del mundo de GO en 2016. El primero utilizaba la potencia de cálculo y bases de datos de partidas. El segundo conseguía aprender a partir de las reglas del juego.
Con estas mismas herramientas, los científicos abordan el objetivo final: desentrañar la estructura de moléculas complejas como las proteínas y conseguir diseñar nuevas estructuras y aplicaciones de estas moléculas tan transcendentales para la vida.
El equipo Hassabis/Jumper entrenó al sistema de inteligencia artificial AlphaFold2 con la enorme información de las bases de datos de todas las estructuras de proteínas y secuencias de aminoácidos conocidas y la nueva arquitectura de IA empezó a dar buenos resultados. Esta versión mejorada logró un hito histórico en el CASP de 2020 –algo así como unas olimpiadas de proteínas–, alcanzando una precisión comparable a la cristalografía de rayos X y resolviendo efectivamente un desafío de 50 años en la predicción de estructuras proteicas.
Antonio M. Rodríguez García, Facultad de Farmacia, Química Orgánica, Universidad de Castilla-La Mancha; Antonio de la Hoz Ayuso, Catedrático de Química Orgánica, Universidad de Castilla-La Mancha y Enrique Díez Barra, Química Orgánica, Universidad de Castilla-La Mancha
Este artículo fue publicado originalmente en The Conversation. Lea el original.